
Ejemplos de imágenes reales de diferentes tipos de células versus imágenes sintéticas producidas por el modelo generativo de los investigadores. CRÉDITO Ali Shariati et al.
Nota del editor: Cuando comencemos a enviar equipos a otros mundos para buscar (y luego cuantificar), las imágenes de formas de vida desempeñarán un papel central. Dada la complejidad de tales incursiones, será esencial integrar capacidades de IA en nuestros compañeros robóticos y en los sensores/tricorders que llevamos. Ante lo totalmente desconocido que enfrentaremos, recurriremos a nuestras herramientas para que nos ayuden a comprender lo que vemos y no solo nos capaciten a nosotros – sino también a nosotros mismos – para comprender lo que nosotros –ellos– vemos.
La observación de células individuales bajo un microscopio puede revelar una variedad de fenómenos biológicos celulares importantes que con frecuencia desempeñan un papel en las enfermedades humanas, pero el proceso de distinguir células individuales entre sí y de sus entornos requiere mucho tiempo y es una tarea muy adecuada para la IA. Asistencia.
Los modelos de IA aprenden a realizar tales tareas utilizando un conjunto de datos anotados por humanos, pero el proceso de distinguir las células de su entorno, llamado “segmentación unicelular”, requiere mucho tiempo y es laborioso. Como resultado, la cantidad de datos anotados para su uso en conjuntos de entrenamiento de IA es limitada. Los investigadores de UC Santa Cruz desarrollaron un método para resolver este problema mediante la construcción de un modelo de IA de generación de imágenes microscópicas para crear imágenes realistas de células individuales, que luego se utilizan como «datos sintéticos» para formar un modelo de IA para realizar mejor la segmentación de células individuales.
El nuevo software se describe en un nuevo artículo publicado en la revista iScience. El proyecto fue dirigido por el profesor asistente de ingeniería biomolecular Ali Shariati y su estudiante de posgrado Abolfazl Zargari. El modelo, llamado cGAN-Seg, está disponible gratis en GitHub.
«Las imágenes de nuestro modelo están listas para usarse para entrenar modelos de segmentación», dijo Shariati. “En cierto sentido, practicamos la microscopía sin microscopio, ya que podemos generar imágenes muy cercanas a las imágenes reales de las células en términos de los detalles morfológicos de cada célula. Lo bueno de todo esto es que cuando salen del modelo, ya están anotados y etiquetados. Las imágenes tienen muchas similitudes con imágenes reales, lo que luego nos permite generar nuevos escenarios que nuestro modelo no vio durante el entrenamiento.
Las imágenes de células individuales vistas con un microscopio pueden ayudar a los científicos a aprender más sobre el comportamiento y la dinámica de las células a lo largo del tiempo, mejorar la detección de enfermedades y encontrar nuevos fármacos. Los detalles subcelulares, como la textura, pueden ayudar a los investigadores a responder preguntas importantes, como si una célula es cancerosa o no.
Sin embargo, encontrar y marcar manualmente los límites de las células a partir de su fondo es extremadamente difícil, especialmente en muestras de tejido donde hay muchas células en una imagen. Los investigadores podrían tardar varios días en realizar manualmente la segmentación celular en sólo 100 imágenes microscópicas.
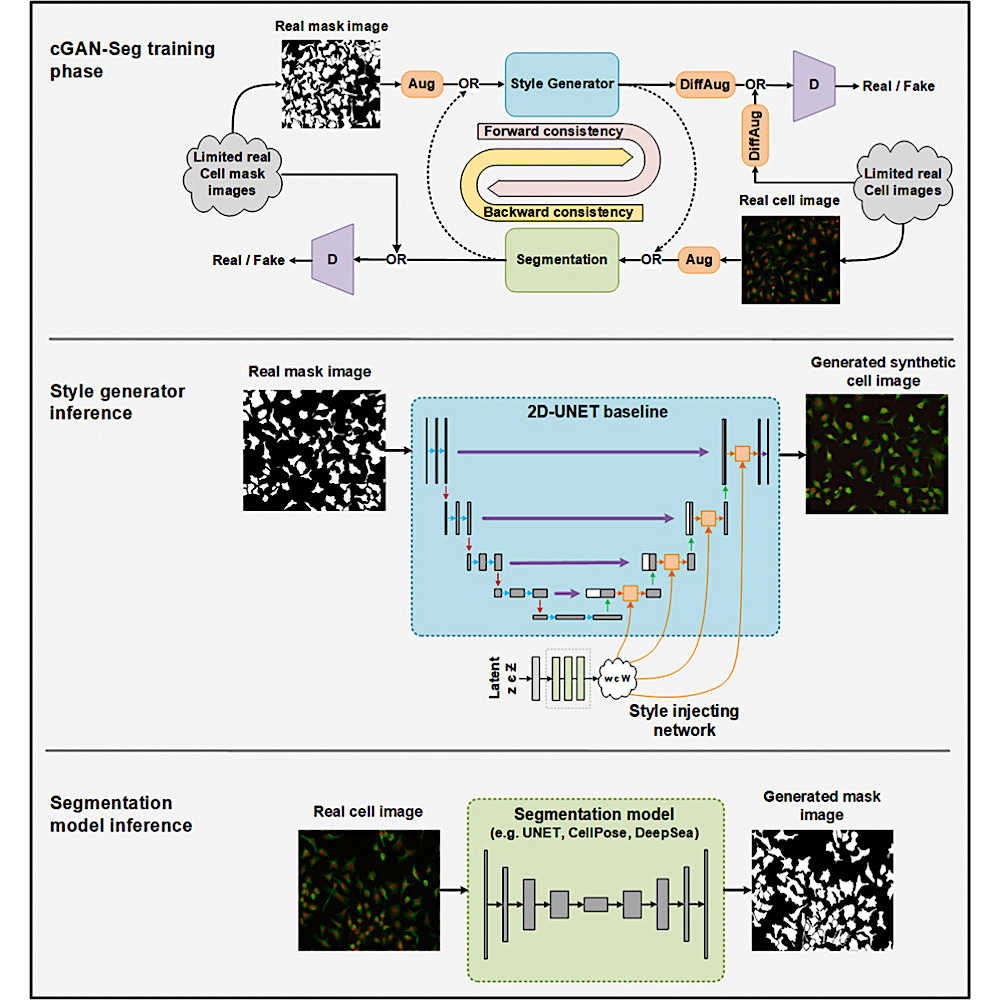
Tres escenarios de entrenamiento de segmentación celular diferentes (A) Entrenamiento de cGAN-Seg en un conjunto de datos limitado de imágenes de células (por ejemplo, 200 muestras de entrenamiento en este artículo) utilizando un diseño personalizado del enfoque CycleGAN de segmentación celular, llamado cGAN-Seg, que incorpora características como la inyección de estilo. , discriminador PatchGan modificado y aumento de imagen diferenciable. (B) Entrenamiento convencional del modelo de segmentación en un conjunto de datos limitado de imágenes celulares. (C) Entrenamiento convencional del modelo de segmentación en un gran conjunto de datos de imágenes celulares (por ejemplo, 1000 muestras de entrenamiento en este documento).
El aprendizaje profundo puede acelerar este proceso, pero se requiere un conjunto inicial de datos de imágenes anotadas para entrenar los modelos: se necesitan al menos miles de imágenes como referencia para entrenar un modelo de aprendizaje profundo preciso. Incluso si los investigadores pudieran encontrar y anotar 1.000 imágenes, es posible que esas imágenes no contengan las variaciones de características que aparecen en diferentes condiciones experimentales.
«Quiere demostrar que su modelo de aprendizaje profundo funciona en diferentes muestras con diferentes tipos de células y diferentes calidades de imagen», dijo Zargari. “Por ejemplo, si entrena su modelo con imágenes de alta calidad, no podrá segmentar imágenes celulares de baja calidad. Rara vez podemos encontrar un conjunto de datos tan poderoso en el campo de la microscopía.
Para abordar este problema, los investigadores crearon un modelo de IA generativa de imagen a imagen que toma un conjunto limitado de imágenes celulares anotadas y etiquetadas y genera más, introduciendo características y estructuras subcelulares más complejas y variadas para crear un conjunto diverso de imágenes «sintéticas». . fotos. En particular, pueden generar imágenes anotadas con una alta densidad de células, particularmente difíciles de anotar a mano y particularmente relevantes para el estudio de tejidos. Esta técnica permite procesar y generar imágenes de diferentes tipos de células, así como diferentes modalidades de imagen, como las tomadas por fluorescencia o tinción histológica.
Zargari, quien dirigió el desarrollo del modelo generativo, utilizó un algoritmo de IA de uso común llamado “red adversaria generativa de ciclo” para crear imágenes realistas. El modelo generativo se ve reforzado por las llamadas «funciones de aumento» y una «red de inyección de estilo», que ayudan al generador a crear una amplia variedad de imágenes sintéticas de alta calidad que muestran diferentes posibilidades en cuanto a la posible apariencia de las células. Hasta donde saben los investigadores, esta es la primera vez que se utilizan técnicas de inyección de estilo en este contexto.
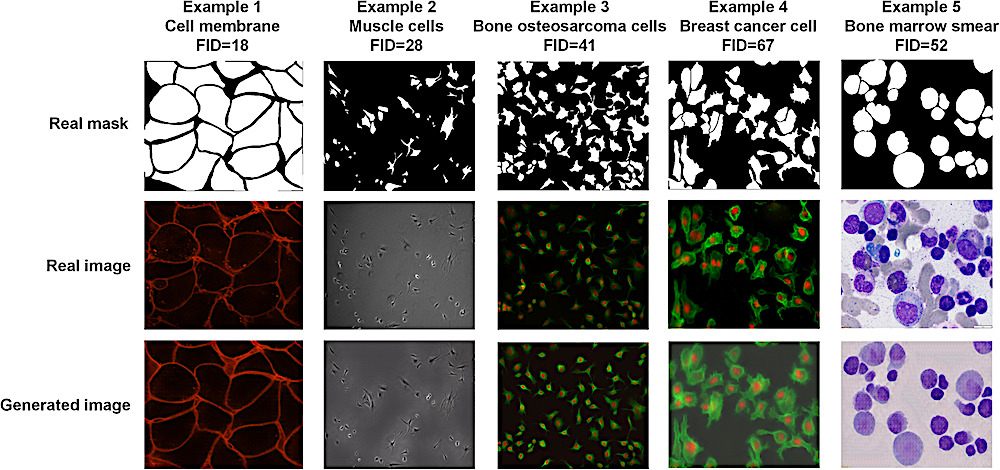
El generador de estilo cGAN-Seg puede generar imágenes sintéticas a través de múltiples modalidades de imágenes, tipos de células y orgánulos subcelulares (de conjuntos de datos DeepSea y CellPose) similares a imágenes reales, como lo muestran las puntuaciones FID relativamente bajas.
Luego, este conjunto diverso de imágenes sintéticas creadas por el generador se utiliza para entrenar un modelo para realizar con precisión la segmentación celular en nuevas imágenes reales tomadas en experimentos.
“Utilizando un conjunto de datos limitado, podemos entrenar un buen modelo generativo. Utilizando este modelo generativo, podemos generar un conjunto más amplio y diverso de imágenes sintéticas anotadas. Utilizando las imágenes sintéticas generadas, podemos entrenar un buen modelo de segmentación; esa es la idea principal”, dijo Zagari.
Los investigadores compararon los resultados de su modelo utilizando datos de entrenamiento sintéticos con métodos más tradicionales de entrenamiento de IA para realizar la segmentación celular entre diferentes tipos de células. Descubrieron que su modelo produce una segmentación significativamente mejorada en comparación con los modelos entrenados con datos de entrenamiento limitados convencionales. Esto confirma a los investigadores que proporcionar un conjunto de datos más diverso al entrenar el modelo de segmentación mejora el rendimiento.
Con estas capacidades de segmentación mejoradas, los investigadores podrán detectar mejor las células y estudiar la variabilidad entre células individuales, particularmente entre células madre. En el futuro, los investigadores esperan utilizar la tecnología que desarrollaron para ir más allá de las imágenes fijas y generar vídeos, lo que puede ayudarles a identificar los factores que influyen en el destino de una célula en las primeras etapas de su vida y a predecir su futuro.
«Generamos imágenes sintéticas que también se pueden transformar en una película de lapso de tiempo, donde podemos generar el futuro invisible de las células», dijo Shariati. «Con esto queremos ver si somos capaces de predecir los estados futuros de una célula, por ejemplo si la célula crecerá, migrará, se diferenciará o se dividirá».
Segmentación celular mejorada con conjuntos de datos de entrenamiento limitados utilizando redes adversarias generadoras de ciclosiScience (acceso gratuito)
Astrobiología
También te puede interesar
-
El rover Perseverance de la NASA en Marte comienza un empinado ascenso hacia el borde del cráter
-
La Tierra se desaceleró significativamente durante la peor extinción masiva jamás registrada, según un estudio dirigido por China
-
Estrellas masivas brillan en esta imagen ultravioleta tomada por el Hubble
-
Astronauta de la NASA comparte foto de la Luna sobre el Pacífico: 'Es impresionante'
-
SpaceX prueba tecnología revolucionaria durante caminata espacial
